单细胞基因组学的快速发展极大推动了我们对组织与器官中细胞异质性的理解。然而,传统的单细胞RNA测序(scRNA-seq)依赖细胞解离处理,导致微环境信息丢失,难以全面刻画细胞间的空间关系。近年来,空间转录组技术的进步使得原位单细胞分析成为可能,能够在多种组织中对数十万细胞的数百个基因进行空间定位。但如何在不断增长的空间数据中提炼跨物种、跨组织的通用细胞表征,仍是一大挑战。
生物医药管线CGT合作大会关注到,为解决这一问题,慕尼黑工业大学与亥姆霍兹慕尼黑研究中心团队于2025年10月30日在《Nature Methods》发表研究,题为“Nicheformer: a foundation model for single-cell and spatial omics”,通讯作者为Fabian J. Theis。团队提出了在大规模单细胞与空间组学数据上预训练的基础模型——Nicheformer,旨在为有限训练样本下的空间任务预测提供统一解决方案。

图片来源:智药邦
为此,他们构建了迄今最大规模的单细胞与空间组学数据集——SpatialCorpus-110M,包含超过1.1亿个细胞。通过引入模态、物种和检测类型的上下文标记,Nicheformer能够实现不同来源数据的联合表示,并在多项下游任务中显著优于现有模型,标志着空间组学计算的基础架构迈入新阶段。
背景
深度学习已成为单细胞生物学的重要工具,通过表示学习实现数据整合、扰动效应预测与图谱映射。近年来,多种基于Transformer架构的单细胞基础模型相继出现,如CellPLM、Geneformer等,依托超大规模细胞数据集进行预训练。然而,这些模型多聚焦于分离单细胞数据,缺乏对空间信息的建模能力。
现有工作中,CellPLM虽考虑了空间关系,但其训练数据仅包括900万分离细胞与200万空间细胞,且主要针对基因插补任务。Nicheformer则首次系统性地在大规模空间与单细胞联合数据上进行预训练,实现跨模态、跨物种、跨任务的统一学习框架。
结合空间与分离单细胞数据的基础模型Nicheformer
图片来源:智药邦
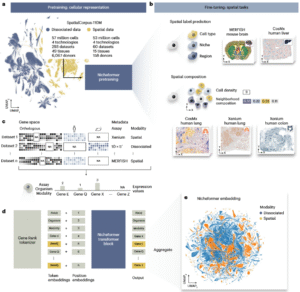
Nicheformer采用Transformer架构,并在SpatialCorpus-110M上完成预训练(图a)。该数据集包含来自分离单细胞与空间测序的超过1.1亿个细胞。模型在两种模态上联合学习,以捕捉跨组织与跨技术的表达模式。
研究团队设定了一系列下游任务,用于验证Nicheformer在空间信息推断与跨模态迁移中的能力(图b)。任务包括预测细胞生态位、组织区域与空间组成等。
此外,作者通过拼接人类与小鼠的同源蛋白编码基因,构建了包含20310个基因token的共享词表(图c)。每个单细胞表达向量被转换为基因排名序列(图d),从而提升模型在批次效应下的鲁棒性。
大规模预训练数据集SpatialCorpus-110M
图片来源:智药邦
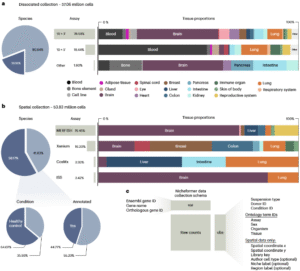
研究团队构建的SpatialCorpus-110M是目前最大的单细胞与空间组学数据集。
其中,分离单细胞部分包含5706万个细胞,来源于17种器官与18种细胞系;空间转录组部分包含5383万个细胞,覆盖15种实体器官,涉及四类空间技术平台(图a,b)。所有数据在基因与细胞层面均进行标准化,采用统一的元数据架构以确保跨模态一致性(图c)。
与性别相关的基因-基因依赖关系
图片来源:智药邦
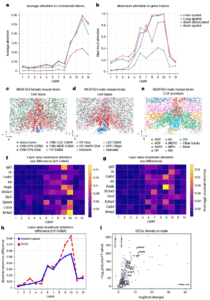
团队对Nicheformer的注意力矩阵进行系统分析,以探索模型是否学会了可泛化的生物结构。结果显示,不同层的注意力呈分层组织结构:早期层较为分散,中间层在特定基因上集中(图b),末层聚焦于上下文标记(图a)。
在雄性与雌性MERFISH小鼠脑数据中,模型注意力显著区分性别特异基因(SDGs)(图c–i)。其中,第九与第十层在HY GABA细胞中表现出最强的性别差异信号,说明Nicheformer能够自动捕捉性别相关的分子模式。
空间标签的跨数据迁移能力
图片来源:智药邦
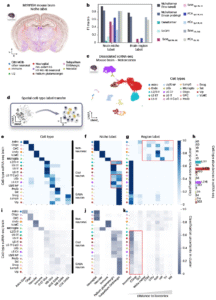
研究团队在MERFISH小鼠脑数据中标注了17个脑区与8个生态位(图a),通过线性探测与微调两种策略评估模型在未见切片上的泛化能力。
结果显示,Nicheformer在宏平均F1分数上超越PCA、scVI及多种基础模型(图b)。
模型还能将MERFISH定义的细胞类型映射至scRNA-seq运动皮层数据中,成功识别出与运动皮层相关的9种细胞类型(图c–i)。预测标签与原始注释高度一致,且分类不确定性较低,显示出强泛化与稳健性。
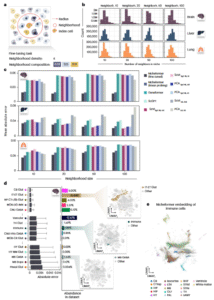
多器官邻域预测性能
图片来源:智药邦
研究者在MERFISH小鼠脑、CosMx人类肝脏与肺部数据集中测试Nicheformer在不同半径下预测细胞邻域组成的能力(图a,b)。
结果显示,无论在10至100邻居范围,基于Nicheformer嵌入的线性探测模型均优于PCA与scVI,仅次于微调模型(图c)。
模型在数量丰富的细胞类型中表现最优,稀有细胞类型预测误差较高(图d),揭示了数据密度对性能的影响。
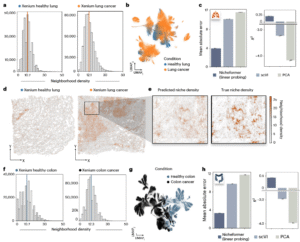
未知数据中的细胞邻域密度推断
图片来源:智药邦
进一步测试显示,Nicheformer可仅基于转录组信息推断组织中细胞的局部密度。在Xenium肺与结肠数据中,模型准确区分出肿瘤与健康区域的密度差异(图a,f)。
与PCA和scVI相比,基于Nicheformer嵌入的线性探测回归模型在MAE与R²指标上表现最佳(图c,h),能够精确识别高密度肿瘤组织(图d,g)。
总结
Nicheformer展示了多尺度基础模型在单细胞与空间组学领域的应用潜力。借助SpatialCorpus-110M,模型不仅能在多任务中稳定泛化,还揭示了空间信息与转录组表达之间的复杂联系。
该研究提出的空间信息迁移框架为解离单细胞数据的环境复原与组织功能预测提供了新路径,也为生物医药管线CGT合作大会关注的精准细胞功能建模与组织重建算法奠定了技术基础。
尽管Nicheformer的表现突出,但仍存在局限:模型性能依赖数据多样性,尚未充分利用物理位置信息,且可解释性有待提升。未来研究需在模型参数规模与预训练深度上继续扩展,以实现更广泛的临床与药物研发应用。
文章来源:智药邦

