生物医药产业大会关注到,昌平实验室赵亚杰团队于11月17日在《核酸研究》(Nucleic Acids Research)发表研究,推出首个复杂性状遗传信号的空间效应分布全景图谱 Spatial GWAS Atlas(https://zhaolab.cpl.ac.cn/spatialgwas)。该研究通过系统整合GWAS与ST数据,建立遗传信号与空间表达模式的映射关系,使复杂性状相关细胞在组织空间维度上实现精细定位,并为疾病遗传机制研究提供新的数据支撑。

图片来源:医药魔方
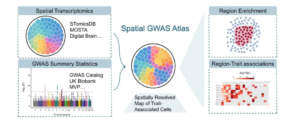
随着基因组测序和人群队列的快速积累,全基因组关联研究(GWAS)已鉴定大量与复杂性状及疾病相关的遗传变异,为疾病机制解析和潜在靶点挖掘带来关键线索。然而,大多数变异位于非编码区,其功能具有高度组织与细胞特异性。空间转录组(ST)技术的出现,使研究者能够在保留空间坐标的条件下进行转录本测序,揭示组织中基因表达的精细空间分布,为理解遗传信号在组织结构中的作用方式提供新路径。现有数据库多仅聚焦单细胞或ST数据,尚未将遗传信号映射至组织空间结构,限制了从组织结构层面理解复杂疾病遗传基础的研究进展。因此,整合GWAS与ST绘制遗传信号空间效应分布图谱具有重要意义。
图片来源:医药魔方
该研究系统整合GWAS与ST数据,建立遗传信号与空间表达模式的映射关系,实现复杂性状相关细胞在组织空间维度的定位。Spatial GWAS Atlas纳入3,854个高质量GWAS数据集与635个ST数据集。GWAS数据来自UK Biobank、FinnGen、Million Veteran Program以及GWAS Catalog等资源。ST数据覆盖多个权威空间组学平台,包括Macaque Spatial Transcriptomics Atlas、Mouse Organogenesis Spatiotemporal Transcriptomics Atlas、Mouse Spatial Transcriptomics Atlas和STOmicsDB,囊括Stereo-seq、10x Visium、MERFISH等主流平台,涉及人类、猕猴、小鼠多个组织类型,如大脑、心脏、肝脏、肺、脾脏、肾脏、睾丸、脊髓、肠道及胚胎等。
针对GWAS与ST数据的异构特性,研究团队设计严格的标准化预处理流程,并构建本体术语驱动的智能匹配系统。通过Ontology Lookup Service与Uberon术语,将GWAS表型与组织类型进行半自动化精准对应,提高关联结果的生物学可解释性。基于gsMap遗传信号空间映射算法,研究团队在单细胞分辨率下系统计算并量化复杂性状(疾病)与空间位点(区域或细胞类型)的关联强度,共获得超过175万条表型–空间区域(或细胞类型)关联数据,构建迄今最全面的复杂性状与疾病的遗传信号空间效应分布图谱。

图片来源:医药魔方
Spatial GWAS Atlas提供浏览、搜索与可视化三大模块。用户可按关键词检索复杂性状、组织或物种,并筛选对应的GWAS与ST数据集。交互式可视化界面展示遗传信号在空间转录组数据中的效应分布,可按注释类型或显著性水平(p值)进行筛选,并支持跨性状、跨物种与跨组织比较分析。
生物医药产业大会关注到,该图谱的构建为解析复杂疾病的遗传基础提供了高维度空间信息支撑。
文章来源:医药魔方


